Chargée de Recherche CNRS
Tel: +33 1-58-41-51-71

Fax: +33 1-58-41-51-74
Courriel: chantal.prevost
 ibpc.fr
ibpc.fr
Piece: 311
Intérêts Scientifiques
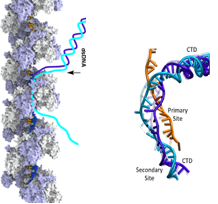
Mécanisme de la recombinaison homologue induite par RecA
Exploration du mécanisme de la reconnaissance de séquence et de l'échange de brins d'ADN au sein du nucléofilament de RecA
Nous avons simulé pour la première fois l'échange d'appariement qui se produit spontanément au sein d'un nucléofilament de RecA lors de la recherche d'homologie de séquence entre brins d'ADN.
- schéma du complexe de recombinaison et du mode possible d'association entre nucléofilament et ADN
- vidéo du modèle que nous proposons pour l'étape initiale de la recombinaison, où 8 nucléotides sont testés par échange très rapide d'appariement (construction et simulation de dynamique moléculaire)
- lire l'article dans Nucleic Acids Res 2015
- visualiser la video des premiers tests de construction du modèle par simulation interactive
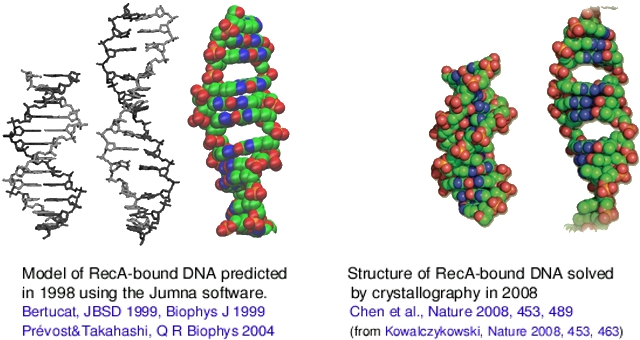
Déformations de grande amplitude de l'ADN
Génération et optimisation d'ADN déformé, notamment par la formation de complexes protéine/ADN
Développement d'algorithmes pour l'amarrage flexible de macromolécules
Dans notre approche, les parties protéiques flexibles sont représentées par des ensembles de conformations (copies) qui couvrent l'espace accessible. Au cours de la simulation d'amarrage, toutes les copies sont prises en compte simultanément, elles sont traitées par la théorie du Champ Moyen.Cette stratégie a été couplée à deux méthodes d'exploration:
- Monte Carlo (amarrage de précision ADN/protéine à résolution atomique) et
- Minimization (amarrage systématique protéine/protéine et protéine/ADN à résolution réduite; Bastard et al., Proteins 2006, 62, 956).
Logiciels
Ptools Une librairie Py/C++ orientée objet qui permet de manipuler et assembler des modèles de protéineset d'ADN en représentation simplifiée (gros grain) ou atomique.
Ptools est à l'origine le support de distribution du programme d'amarrage ATTRACT ( M. Zacharias)
Ptools a été conçu et développé par Adrien Saladin
Il est maintenu et développé par Benoist Laurent, Charles Robert, Pierre Poulain et Chantal Prévost
PTools est librement accessible sur Github https://github.com/ptools/ptools/tree/develop
MC2 Une approche par Monte Carlo / Multi Copie pour l'amarrage flexible de précision
protéine/ADN
par Karine Bastard, Aurélien Thureau et Chantal Prévost
Ateliers et Projets
► CECAM Workshop "Flexible Docking", Lyon France (2004)► ACI IMPBio : projet ModRecHom (2004-2007)
"Modélisation multi-échelle d'interactions moléculaires dynamiques. Application à la recombinaison"
en collaboration avec Jean-Louis Viovy, Institut Curie Paris et Marie Dutreix, Institut Curie Orsay
